Emotion-Antecedent Appraisal Checks : EEG and EMG data sets for Novelty and Pleasantness
이 데이터 세트는 van Peer et al에 의해 구성되었으며 https://zenodo.org/record/197404#.XZtCzy2B0UE에서 다운로드할 수 있다 . (van Peer, Jacobien M., Coutinho, Eduardo, Grandjean, Didier, & Scherer, Klaus R. (2017). 감정 선행 평가 검사 : EEG 및 EMG 데이터 세트 for Novelty and Pleasantness[데이터 세트]. PloS One. Zenodo. http://doi.org/10.5281/zenodo.197404)
# For elimiating warnings
from warnings import simplefilter
# ignore all future warnings
simplefilter(action='ignore', category=FutureWarning)
import numpy as np
import mne
from mne.io import concatenate_raws, read_raw_fif
import mne.viz
from os import walk
참가자 9에 속한 파일 가져오기
files = []
path = '../../study1/study1_eeg/'
participant_prefix = 'P-09_'
for (dirpath, dirnames, filenames) in walk(path):
new_names = [dirpath+f for f in filenames if (participant_prefix in f)]
files.extend(new_names)
break
CSV 파일을 NumPy 배열로 읽기
tmp = np.loadtxt(files[0], delimiter=',')
n_channels = tmp.shape[0]
n_times = tmp.shape[1]
participant_data = np.ndarray((len(files),n_channels,n_times))
for trial in range(0,len(files)):
new_data = np.loadtxt(files[trial], delimiter=',')
if trial == 0:
print('n_channels, n_times: ' + str(new_data.shape))
new_data = new_data.astype(float)
participant_data[trial] = new_data
print('Number of epochs: ' + str(participant_data.shape))n_channels, n_times: (64, 384)
Number of epochs: (380, 64, 384)
파일 이름에서 이벤트 이름 추출
epochs_events = []
for f in files:
res = f.split('_')
epochs_events.append(res[-2])
epochs 객체의 이벤트 매개변수는 (n_epochs,3)과 같은 차원을 갖는 numpy ndarray이다. 각 epoch에 대해 다음과 같은 구조를 갖는다. (event_sample, previous_event_id, event_id)
unique_events = list(set(epochs_events))
print(unique_events)
unique_events = sorted(unique_events)
print(unique_events)
unique_events_num = [i for i in range(len(unique_events))]
epoch_events_num = np.ndarray((len(epochs_events),3),int)
for i in range(len(epochs_events)):
for j in range(len(unique_events)):
if epochs_events[i] == unique_events[j]:
epoch_events_num[i,2] = unique_events_num[j]
if i >0:
epoch_events_num[i,1] = epoch_events_num[i-1,2]
else:
epoch_events_num[i,1] = unique_events_num[j]
epoch_events_num[i,0] = i
event_id = {}
for i in range(len(unique_events)):
event_id[unique_events[i]] = unique_events_num[i]
print(event_id)['NU', 'FN', 'FU', 'FP', 'NP', 'NN']
['FN', 'FP', 'FU', 'NN', 'NP', 'NU']
{'FN': 0, 'FP': 1, 'FU': 2, 'NN': 3, 'NP': 4, 'NU': 5}
채널의 위치를 가져온다. 데이터는 64개 채널이 있는 바이오세미 eeg 장치로 수집된다. 따라서 채널 위치를 얻으려면 biosemi64를 read_montage 함수에 전달해야 한다.
%matplotlib inline
montage = mne.channels.read_montage('biosemi64')
print('Number of channels: ' + str(len(montage.ch_names)))
montage.plot(show_names=True)Number of channels: 67
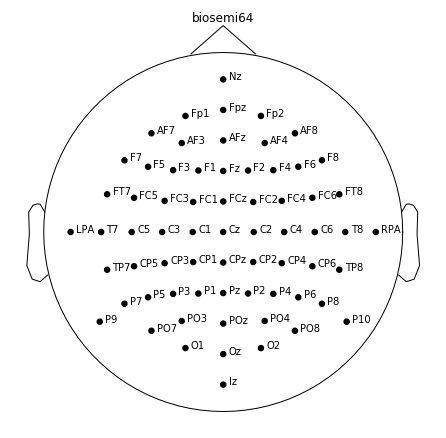
몽타주는 64+3개의 채널을 가지고 있지만 데이터는 동일한 장치로 수집되지만 데이터는 64개의 채널이 있음을 유의해야 한다. 추가 3개의 채널은 기준점이며 참조용으로 존재한다. 데이터와 motang을 일치시키기 위해 fudicials를 제거해야 한다.
n_channels = 64
fiducials = ['Nz', 'LPA', 'RPA']
ch_names = montage.ch_names
ch_names = [x for x in ch_names if x not in fiducials]
print('Number of cahnnels after removing the fudicials: '+ str(len(ch_names)))
# Specify ampling rate
sfreq = 256 # HzNumber of cahnnels after removing the fudicials: 64
epochs 객체에 대한 정보 구조를 만든 다음 참가자의 데이터, info 객체 및 이벤트를 유지하는 numpy 배열로 epochs 객체를 만든다.
epochs_info = mne.create_info(ch_names, sfreq, ch_types='eeg')
epochs = mne.EpochsArray(data=participant_data, info=epochs_info, events=epoch_events_num, event_id=event_id)
epochs.set_montage(montage)
epochs.drop_bad()380 matching events found
No baseline correction applied
Not setting metadata
0 projection items activated
0 bad epochs dropped
플롯 에포크, 에포크 및 이벤트의 평균
# Plot of averaged epochs
epochs.average().plot()
#plot events
mne.viz.plot_events(epochs.events)
# Plot of epochs
%matplotlib tk
mne.viz.plot_epochs(epochs, scalings='auto')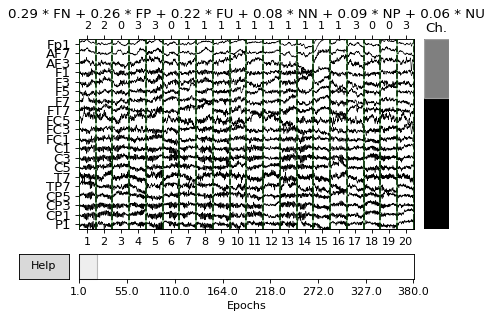
에포크를 확장자 .fif인 파일에 저장한다.
epochs.save('../../study1/study1_eeg/epochdata/'+participant_prefix[:-1]+'.fif', verbose='error')
https://neuro.inf.unibe.ch/AlgorithmsNeuroscience/Tutorial_files/DatasetConstruction.html
'Brain Engineering > MNE' 카테고리의 다른 글
| Machine Learning : Single-Participant Analysis (2) (0) | 2022.04.05 |
|---|---|
| Machine Learning : Single-Participant Analysis (1) (0) | 2022.04.05 |
| Preprocessing : Choosing a Reference (0) | 2022.04.05 |
| Preprocessing : Baseline Correction (0) | 2022.04.05 |
| Preprocessing : Data Visualization (0) | 2022.04.05 |



