728x90
반응형
SMALL
Group-Level Analysis on Temporal Dimension
Group-Level Analysis에서와 같이 참가자의 데이터를 연결하여 그룹 수준의 분석에 필요한 데이터 세트를 구성한다.
#Load Dataset
data_folder = '../../study1/study1_eeg/epochdata/'
files = [data_folder+f for f in listdir(data_folder) if isfile(join(data_folder, f)) and '.DS_Store' not in f]
#files = files[:10]
ids = [int(f[-6:-4]) for f in files]
numberOfEpochs = np.zeros((len(ids), 3))
# Read the EEG epochs:
epochs_all_UN, epochs_all_UP, epochs_all_NP = [], [], []
for f in range(len(files)):
epochs = mne.read_epochs(files[f], verbose='error')
epochs_UN = epochs['FU', 'FN']
epochs_UP = epochs['FU', 'FP']
epochs_NP = epochs['FN', 'FP']
numberOfEpochs[f,0] = int(len(epochs_UN.events))
numberOfEpochs[f,1] = int(len(epochs_UP.events))
numberOfEpochs[f,2] = int(len(epochs_NP.events))
UN, UP, NP = [ids[f]], [ids[f]], [ids[f]]
UN.append(epochs_UN)
UP.append(epochs_UP)
NP.append(epochs_NP)
epochs_all_UN.append(UN)
epochs_all_UP.append(UP)
epochs_all_NP.append(NP)
epochs_all_UN = np.array(epochs_all_UN)
epochs_all_UP = np.array(epochs_all_UP)
epochs_all_NP = np.array(epochs_all_NP)
데이터 세트를 얻은 후 데이터, 레이블 및 id (참가자 ID)는 다음 함수로 구분된다.
def getData_labels(epochs):
data, labels, ids = [], [], []
for p in epochs:
tmp_epoch = p[1]
tmp_labels = tmp_epoch.events[:,-1]
labels.extend(tmp_labels)
tmp_id = p[0]
ids.extend([tmp_id]*len(tmp_labels))
data.extend(tmp_epoch.get_data())
data = np.array(data)
labels = np.array(labels)
ids = np.array(ids)
return data, labels, ids
Task #1 : Classification of Unpleasant and Pleasant Events
Python의 sklearn 패키지는 모델을 빌드하는 동안 해당 값을 처리할 수 없기 때문에 모델 구축 단계를 시작하기 전에 데이터에 더하기 / 빼기 무한 또는 NaN이 될 수 있는 잘못된 값이 포함되어 있는지 확인해야 한다. 다음 셀이 "Nan or infinite is detedted!"를 반환하면 다음 단계로 이동하기 전에 데이터 세트를 확인해야 한다. 데이터에 NaN 또는 무한 값이 포함되지 않은 경우가 발생할 수 있지만 sklearn은 여전히 반대 주장에 오류가 발생할 수 있다. 이 오류의 이유는 데이터 세트에 float64 또는 float32 (처음에 데이터를 읽는 동안 선호도에 따라 다름)가 처리할 수 있는 것보다 정밀도가 더 높은 일부 값이 있기 때문이다. 이 문제를 해결하려면 데이터 세트를 읽는 동안 값을 float64 또는 float32로 캐스트할 수 있다.
#Preparing dataset
data_UP, labels_UP, ids_UP = getData_labels(epochs_all_UP)
if np.isfinite(data_UP).all() == True and np.isnan(data_UP).any() == False:
print('Data does not contain nan or infinite value')
else:
print('Nan or inifininte is detected!')Data does not contain nan or infinite valueclf_UP = make_pipeline(Vectorizer(), StandardScaler(), LinearDiscriminantAnalysis(solver='svd'))
sl_UP = SlidingEstimator(clf_UP, scoring='accuracy')
if np.isfinite(data_UP).all() == True and np.isnan(data_UP).any() == False:
CV_score_time = cross_val_multiscore(sl_UP, data_UP, labels_UP, cv=3)
print('Cross validation scores:\n {}'.format(CV_score_time))
else:
print('Input contains NaN or infinity!')[........................................] 100.00% Fitting SlidingEstimator |
[........................................] 100.00% Fitting SlidingEstimator |
[........................................] 100.00% Fitting SlidingEstimator |
Cross validation scores:
[[0.53310105 0.51881533 0.52473868 ... 0.50557491 0.50383275 0.50627178]
[0.51672474 0.50243902 0.50696864 ... 0.49512195 0.49198606 0.48954704]
[0.51672474 0.51881533 0.5097561 ... 0.51184669 0.51010453 0.50383275]]plotCVScores(epochs_UP.times, CV_score_time)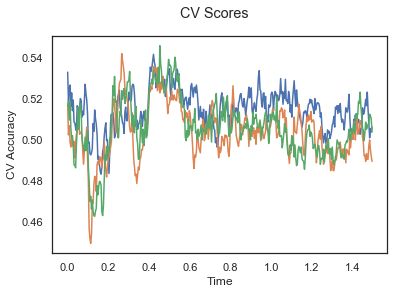
Task #2 : Classification of Unpleasant and Neutral Events
#Preparing dataset
data_UN, labels_UN, ids_UN = getData_labels(epochs_all_UN)
하이퍼파라미터 최적화는 계산 시간 제약으로 인해 적용되지 않는다.
clf_UN = make_pipeline(Vectorizer(), StandardScaler(), LinearDiscriminantAnalysis(solver='svd'))
sl_UN = SlidingEstimator(clf_UN, scoring='accuracy')
if np.isfinite(data_UN).all() == True and np.isnan(data_UN).any() == False:
CV_score_time = cross_val_multiscore(sl_UN, data_UN, labels_UN, cv=3)
print('Cross validation scores:\n {}'.format(CV_score_time))
plotCVScores(epochs_UN.times, CV_score_time)
else:
print('Input contains NaN or infinity!')[........................................] 100.00% Fitting SlidingEstimator |
[........................................] 100.00% Fitting SlidingEstimator |
[........................................] 100.00% Fitting SlidingEstimator |
Cross validation scores:
[[0.50926898 0.51031829 0.50996852 ... 0.51626443 0.51766352 0.51416579]
[0.49825052 0.5003499 0.50909727 ... 0.50979706 0.50524843 0.50559832]
[0.50104969 0.50524843 0.50804759 ... 0.50244927 0.49230231 0.49580126]]
Task #3 : Classification of Neutral and Pleasant Events
#Preparing dataset
data_NP, labels_NP, ids_NP = getData_labels(epochs_all_NP)
clf_NP = make_pipeline(Vectorizer(), StandardScaler(), LinearDiscriminantAnalysis(solver='svd'))
sl_NP = SlidingEstimator(clf_NP, scoring='accuracy')
if np.isfinite(data_NP).all() == True and np.isnan(data_NP).any() == False:
CV_score_time = cross_val_multiscore(sl_NP, data_NP, labels_NP, cv=3)
print('Cross validation scores:\n {}'.format(CV_score_time))
plotCVScores(epochs_NP.times, CV_score_time)
else:
print('Input contains NaN or infinity!')[........................................] 100.00% Fitting SlidingEstimator |
[........................................] 100.00% Fitting SlidingEstimator |
[........................................] 100.00% Fitting SlidingEstimator |
Cross validation scores:
[[0.50223906 0.50189459 0.49362728 ... 0.49741647 0.50912849 0.50740613]
[0.50034459 0.5103377 0.50964852 ... 0.50482426 0.51171606 0.50999311]
[0.49689869 0.50379049 0.51343901 ... 0.50241213 0.51171606 0.49758787]]
위의 세 가지 플롯은 각 분류 작업에 대한 시간 경과에 따른 평균 교차 검증 정확도를 보여준다. 정확도는 모든 시점에 대해 50% 미만이므로 모델이 확률 이상으로 수행되지 않는다. 그룹 수준 교차 검증 플롯과 달리 시간 경과에 따른 모든 참가자의 교차 검증 점수 평균은 50%보다 높다. 따라서 참가자 간의 편차가 크면 그룹 수준에서 성능이 저하된다는 결론을 내릴 수 있다.
Classication over time
Classication over time Tutorial #6: Applying Machine Learning Methods to Temporal Dimensions of EEG Data In this tutorial, we will apply classific...
neuro.inf.unibe.ch
728x90
반응형
LIST
'Brain Engineering > MNE' 카테고리의 다른 글
| [MNE-Python] 선형회귀를 이용하여 EEG 신호에서 안구 운동 제거 (0) | 2022.08.23 |
|---|---|
| 전력 스펙트럼 밀도 (Power Spectral Density) (0) | 2022.08.06 |
| Machine Learning : Classication over time (1) (0) | 2022.04.05 |
| Machine Learning : Group-Level Analysis (Part - 2) (3) (0) | 2022.04.05 |
| Machine Learning : Group-Level Analysis (Part - 1) (2) (0) | 2022.04.05 |



