SVM (Support Vector Machine)
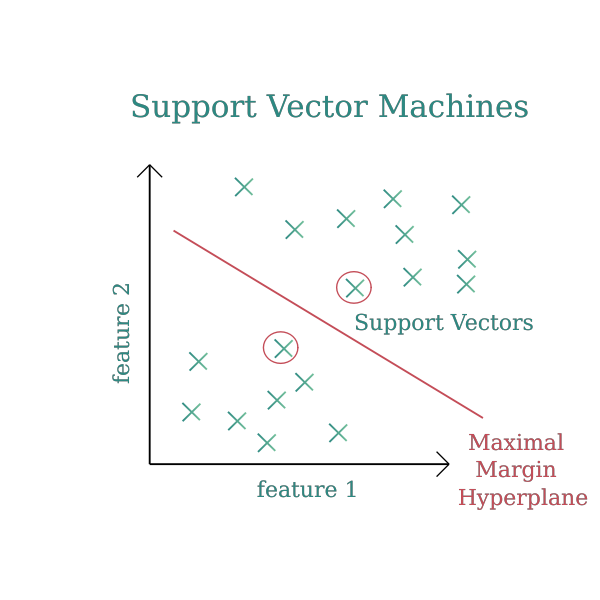
선형이나 비선형 분류, 회귀, 이상치 탐색 등에 사용할 수 있는 다목적 머신러닝 모델이다. 2개의 그룹을 분류하는 가장 일반화된 경계선을 찾기 위한 수학 이론에 의해 발전된 알고리즘이다. 예를 들어, 국경선 결정 문제, 양 국가의 불만을 최소화할 수 있는 최대 거리로 설정한다. 그리고 복잡한 패턴의 문제를 해결하기 위해 주로 사용한다. 중소형 크기의 데이터셋에 적합하다.
SVM은 클래스 사이에 가장 폭이 넓은 도로를 찾는 것과 같다. 이에 SVM를 large margin classification 이라고도 한다. 분류 결정 경계는 도로 경계에 위치한 샘플에 전적으로 결정된다. 이러한 샘플을 support vector라고 한다 (동그라미 데이터). SVM Classifier는 클래스를 나누는 직선 생성, 직선과 가장 가까운 각 클래스의 인스턴스의 거리 (Margin)가 같고, 최대가 되어야 한다.
하드 마진 분류 (Hard Margin Classification)
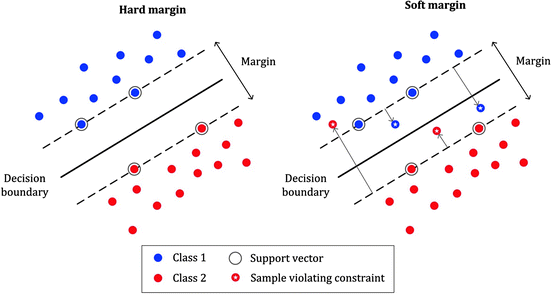
모든 샘플이 도로 바깥쪽에 올바르게 분류되어 있을 때 사용한다. 문제점은 데이터가 선형적으로 구분될 수 있어야 잘 작동하며, 이상치에 민감하다.
소프트 마진 분류 (Soft Margin Classification)
도로의 폭을 가능한 넓게 유지하는 것과 마진 오류 (margin violation) 사이에 적절한 균형을 갖춘 유연한 모델이다.
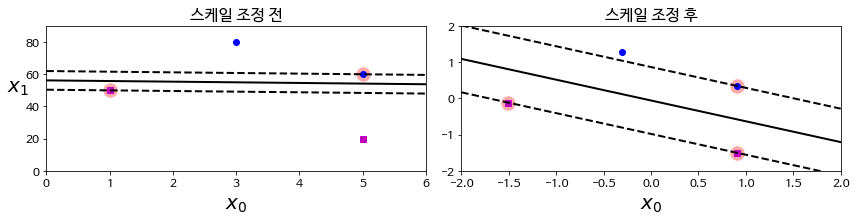
SVM은 특성의 스케일에 민감함하다. 왼쪽 그래프는 수직축 (X1)의 스케일 (0~80)이 수평축 (X0)의 스케일 (0~6)보다 훨씬 커서 가장 넓은 도로가 거의 수평이 된다. 동일한 데이터를 양축에 동일한 값들로 스케일을 조정하면 오른쪽 그래프와 같이 폭이 넓은 도로를 찾을 수 있다.
Linear SVM
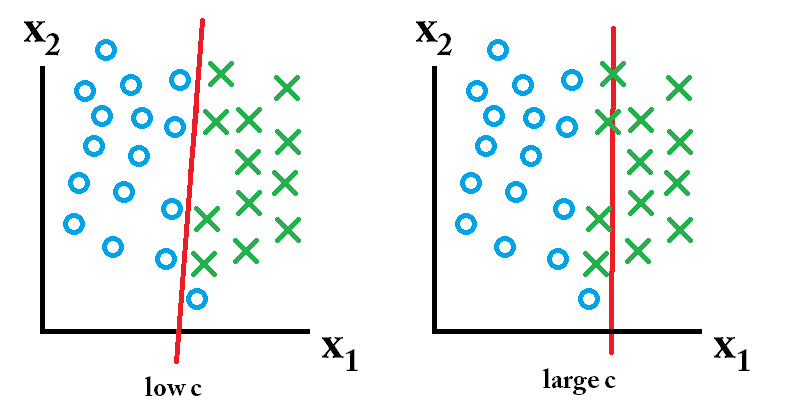
Regularization parameter C를 통해 조절한다. 이 값이 높으면 과대적합, 작으면 과소적합이 일어 난다. 왼쪽 모델이 마진 오류가 많지만 일반화가 더 잘 된 상태이다. SVM 모델이 과대적합 상태가 되면 C 파라미터를 감소시켜 규제를 진행한다.
import numpy as np
from sklearn import datasets
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.svm import LinearSVC
iris = datasets.load_iris()
X=iris['data'][:,(2,3)]
y=(iris['target']==2).astype(np.float64)
svm_clf = Pipeline([
('scaler',StandardScaler()),
('linear_svc',LinearSVC(C=1,loss='hinge'))
])
svm_clf.fit(X,y)import matplotlib.pyplot as plt
X_test_1 = np.arange(1,7,0.1)
X_test_2 = np.arange(0.1,2.5,0.1)
X_test = []
for i in X_test_1:
for j in X_test_2:
X_test.append([i,j])
plt.figure(figsize=(6,8))
for data in X_test:
predict_class = svm_clf.predict([data])
if predict_class[0] == 0:
plt.plot(data[0],data[1],'bo') # Virginia
else:
plt.plot(data[0],data[1],'ro') # Not Virginia
plt.xlabel('Length')
plt.ylabel('Width')
plt.show()
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
iris = sns.load_dataset('iris')sns.pairplot(iris, hue = 'species', palette = 'Dark2')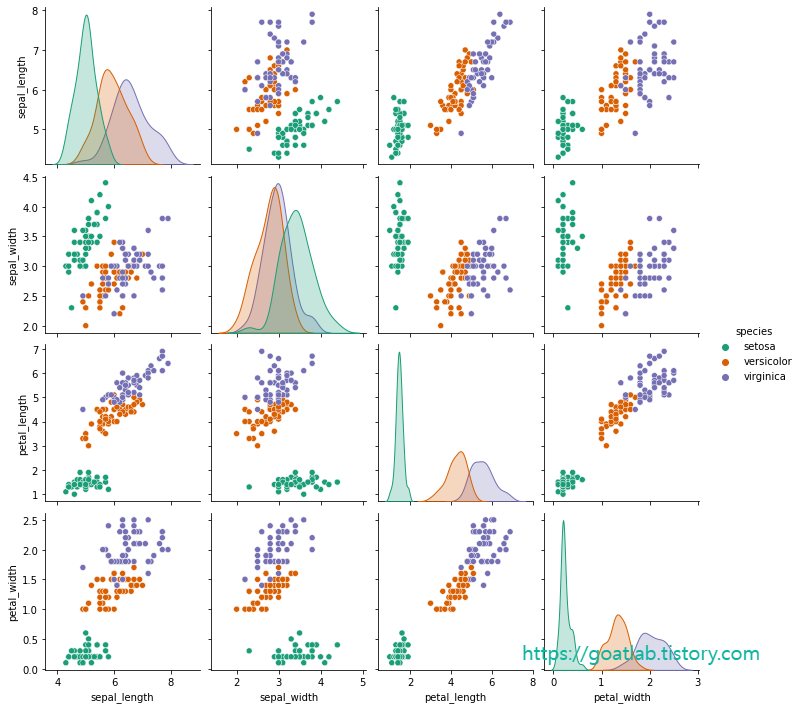
from sklearn.model_selection import train_test_split
from sklearn.metrics import classification_report,confusion_matrix
X = iris.drop('species',axis=1)
y = iris['species']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.30)from sklearn.svm import SVC
svc_model = SVC()svc_model.fit(X_train,y_train)
predictions = svc_model.predict(X_test)print(confusion_matrix(y_test, predictions))print(classification_report(y_test, predictions))grid_predictions = grid.predict(X_test)
print(confusion_matrix(y_test, grid_predictions))print(classification_report(y_test, grid_predictions))from sklearn.model_selection import GridSearchCV
param_grid = {'C': [0.1, 1, 10, 100], 'gamma': [1, 0.1, 0.01, 0.001]}
grid = GridSearchCV(SVC(), param_grid, refit = True, verbose = 2)
grid.fit(X_train, y_train)import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
sns.set_style('whitegrid')from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
col_names = list(cancer.feature_names)
col_names.append('target')
df = pd.DataFrame(np.c_[cancer.data, cancer.target], columns=col_names)
df.head(3)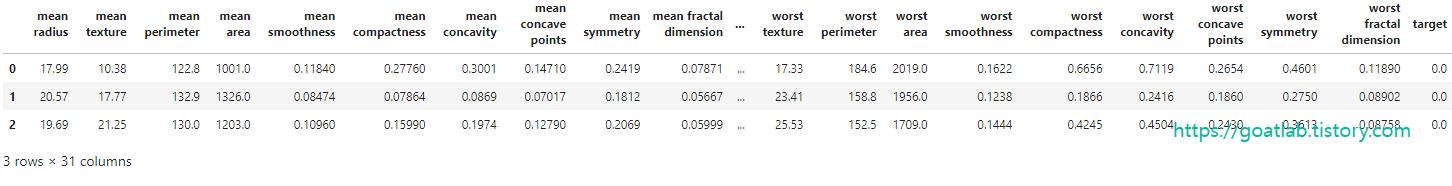
(cancer.target_names)array(['malignant', 'benign'], dtype='<U9')df.columnsIndex(['mean radius', 'mean texture', 'mean perimeter', 'mean area',
'mean smoothness', 'mean compactness', 'mean concavity',
'mean concave points', 'mean symmetry', 'mean fractal dimension',
'radius error', 'texture error', 'perimeter error', 'area error',
'smoothness error', 'compactness error', 'concavity error',
'concave points error', 'symmetry error', 'fractal dimension error',
'worst radius', 'worst texture', 'worst perimeter', 'worst area',
'worst smoothness', 'worst compactness', 'worst concavity',
'worst concave points', 'worst symmetry', 'worst fractal dimension',
'target'],
dtype='object')sns.pairplot(df, hue='target',
vars = ['mean radius', 'mean texture', 'mean perimeter', 'mean area', 'mean smoothness', 'mean compactness','mean concavity', 'mean concave points', 'mean symmetry', 'mean fractal dimension'])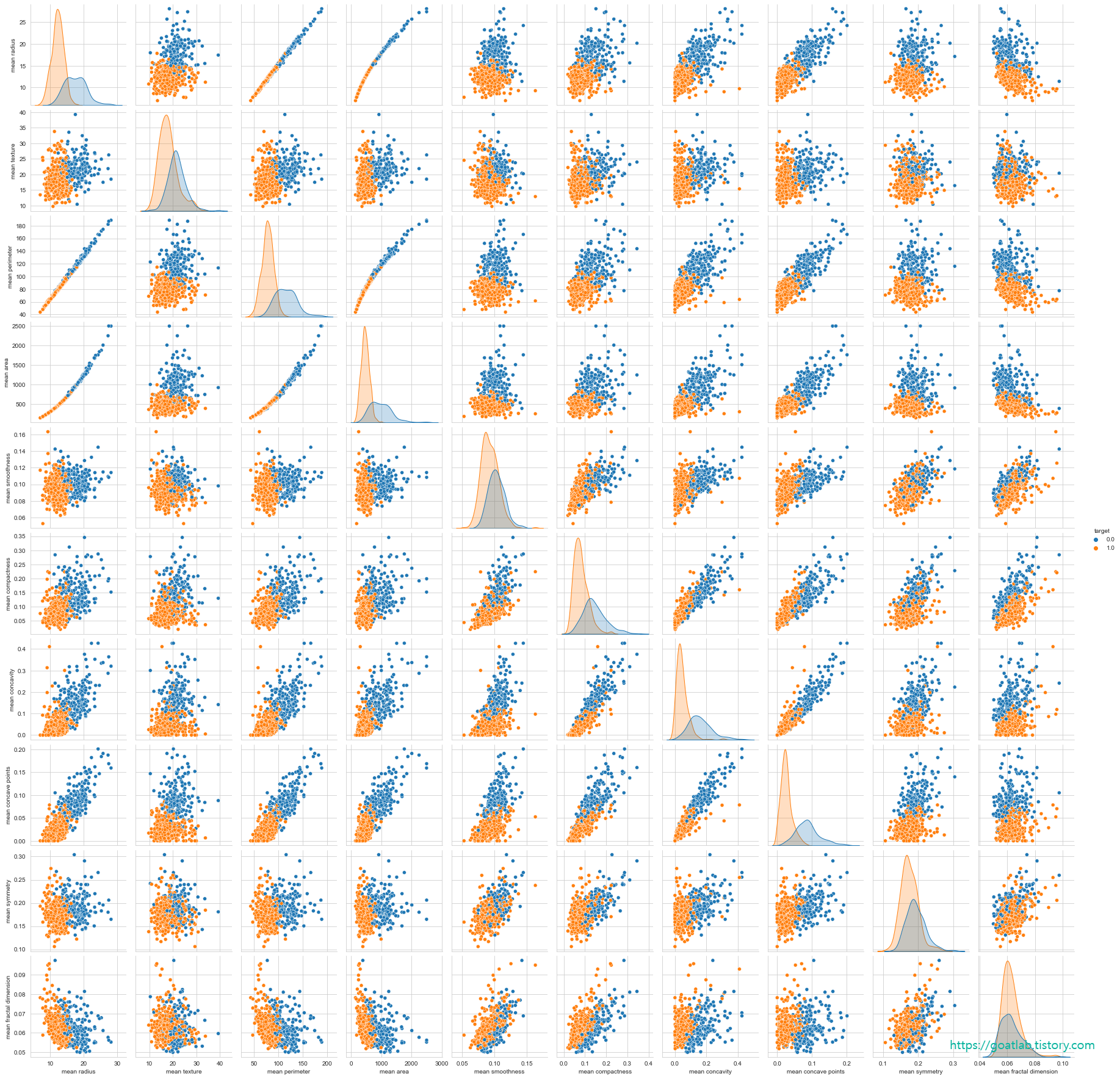
plt.figure(figsize=(20,10))
sns.heatmap(df.corr(), annot=True)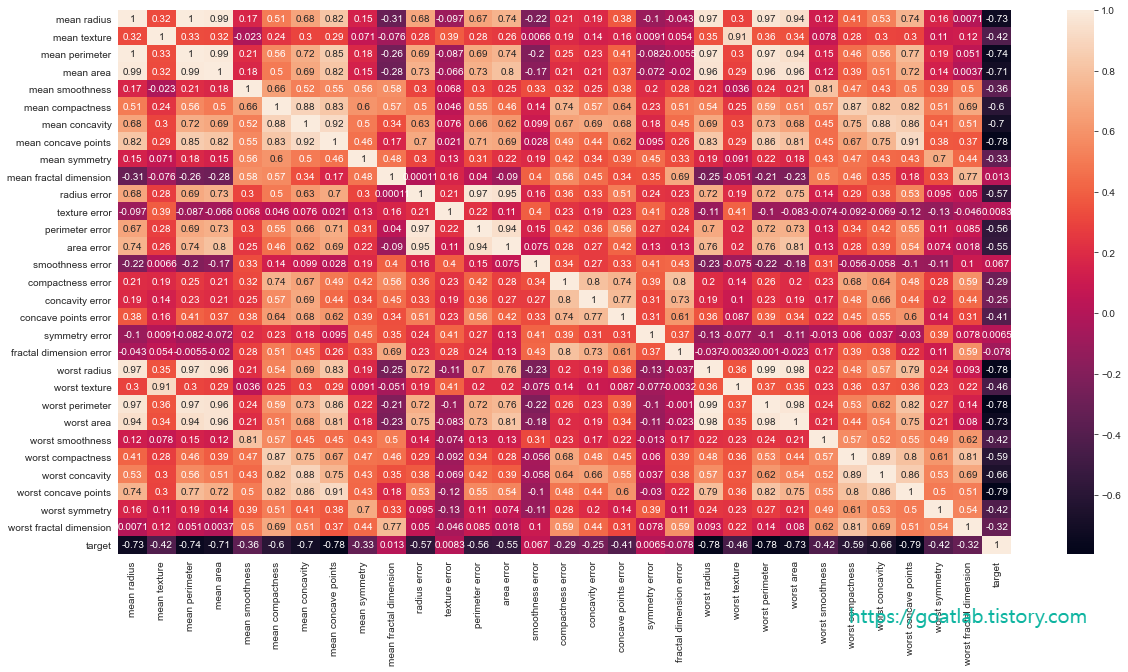
'Learning-driven Methodology > ML (Machine Learning)' 카테고리의 다른 글
| 09. 주성분 분석 (PCA)과 밀도기반 군집분석 (DBSCAN) (0) | 2021.12.15 |
|---|---|
| 08. 텍스트 마이닝 (Text mining) (0) | 2021.12.15 |
| 06. 나이브 베이즈 (Naive Bayes) (0) | 2021.12.15 |
| 06. KNN (K-Nearest Neighbor) (0) | 2021.12.08 |
| 04. 군집 분석 (Clustering) (0) | 2021.12.08 |



