import os
import numpy as np
import matplotlib.pyplot as plt
# the following import is required for matplotlib < 3.2:
from mpl_toolkits.mplot3d import Axes3D # noqa
import mne
sample_data_folder = mne.datasets.sample.data_path()
sample_data_raw_file = os.path.join(sample_data_folder, 'MEG', 'sample',
'sample_audvis_raw.fif')
raw = mne.io.read_raw_fif(sample_data_raw_file, preload=True, verbose=False)
About montages and layouts
Montages은 기존 EEG / MEG 데이터에 할당할 수 있는 3D 센서 위치 (미터의 x, y, z)를 포함한다. 뇌에 상대적인 센서의 위치를 지정함으로써 Montages 순방향 솔루션과 역 추정값을 계산하는 데 중요한 역할을 한다.
대조적으로, 센서 위치의 이상적인 2D 표현 Layouts이 있다. 이것은 주로 토포플롯에 개별 센서 서브플롯을 배열하거나 위에서 본 센서 의 대략적인 상대적 배열을 표시하는 데 사용된다.
Working with built-in montages
MEG 센서의 3D 좌표는 MEG 시스템의 원시 녹음에 포함된다. 로드시 객체의 info 속성에 자동으로 저장된다. Raw EEG 전극 위치는 머리 모양의 차이로 인해 훨씬 더 다양하다. 많은 EEG 시스템에 대한 이상적인 몽타주가 MNE-Python에 포함되어 있다. 이 파일은 폴더 의 mne-python 디렉토리에 저장된다.
montage_dir = os.path.join(os.path.dirname(mne.__file__),
'channels', 'data', 'montages')
print('\nBUILT-IN MONTAGE FILES')
print('======================')
print(sorted(os.listdir(montage_dir)))
---
BUILT-IN MONTAGE FILES
======================
['EGI_256.csd', 'GSN-HydroCel-128.sfp', 'GSN-HydroCel-129.sfp', 'GSN-HydroCel-256.sfp', 'GSN-HydroCel-257.sfp', 'GSN-HydroCel-32.sfp', 'GSN-HydroCel-64_1.0.sfp', 'GSN-HydroCel-65_1.0.sfp', 'artinis-brite23.elc', 'artinis-octamon.elc', 'biosemi128.txt', 'biosemi16.txt', 'biosemi160.txt', 'biosemi256.txt', 'biosemi32.txt', 'biosemi64.txt', 'easycap-M1.txt', 'easycap-M10.txt', 'mgh60.elc', 'mgh70.elc', 'standard_1005.elc', 'standard_1020.elc', 'standard_alphabetic.elc', 'standard_postfixed.elc', 'standard_prefixed.elc', 'standard_primed.elc']
이러한 내장 EEG 몽타주는 다음과 같이 로드할 수 있다 (확장자 없이mne.channels.make_standard_montage 파일 이름을 제공해야 함).
ten_twenty_montage = mne.channels.make_standard_montage('standard_1020')
print(ten_twenty_montage)
Out: <DigMontage | 0 extras (headshape), 0 HPIs, 3 fiducials, 94 channels>
로드되면 몽타주가 set_montage 메서드를 사용하여 데이터에 적용할 수 있다 (ex. raw.set_montage, set_montage 파일 이름 문자열을 메서드에 직접 전달하여 로드 단계를 건너뛸 수도 있다). 채널 이름이 표준 10 – 20 몽타주에 있는 채널 이름과 일치하지 않기 때문에 샘플 데이터에서는 작동하지 않는다. 따라서 여기서는 다음 명령을 실행하지 않는다.
# these will be equivalent:
# raw_1020 = raw.copy().set_montage(ten_twenty_montage)
# raw_1020 = raw.copy().set_montage('standard_1020')
Montage 객체에는 plot2D 또는 3D로 센서 위치를 시각화하는 방법이 있다.
fig = ten_twenty_montage.plot(kind='3d')
fig.gca().view_init(azim=70, elev=15) # set view angle
ten_twenty_montage.plot(kind='topomap', show_names=False)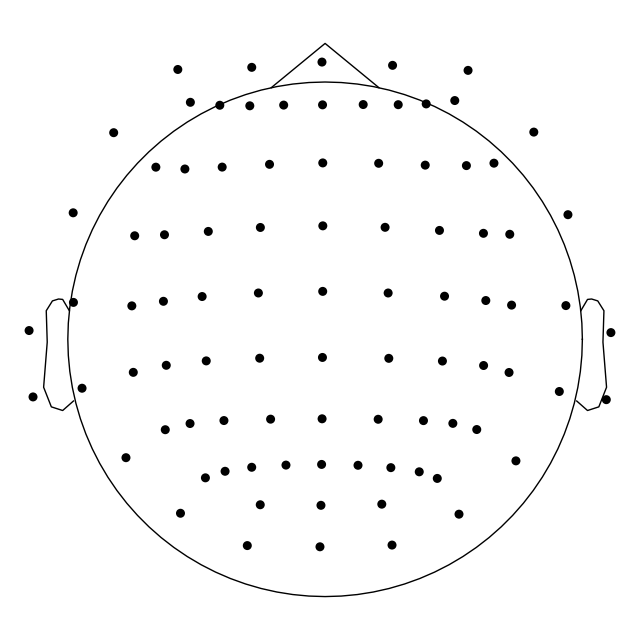
Controlling channel projection (MNE vs EEGLAB)
2D 공간의 채널 위치는 실제 3D 위치를 구에 투영한 다음 구를 평면에 투영하여 얻는다. 몽타주는 (이상화된 구형과 반대되는) 사실적인 채널 위치를 포함 하기 때문에 'standard_1020' 다른 몽타주를 사용하여 채널이 2D로 투영되는 방법을 보여준다.
biosemi_montage = mne.channels.make_standard_montage('biosemi64')
biosemi_montage.plot(show_names=False)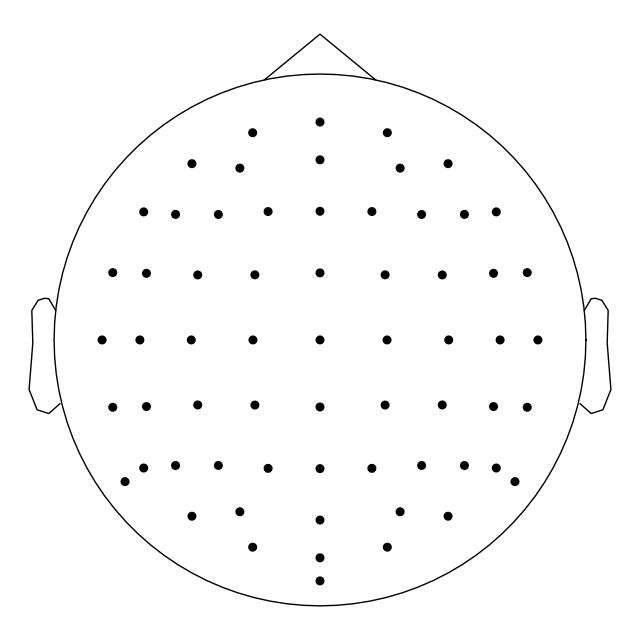
---
Creating RawArray with float64 data, n_channels=64, n_times=1
Range : 0 ... 0 = 0.000 ... 0.000 secs
Ready.
기본적으로 원점이 (x, y, z 좌표)이고 반경이 미터 (9.5cm)인 구가 사용된다. 채널을 2D로 플롯하는 모든 함수에서 단일 값을 인수로 전달하여 다른 구 반경을 사용할 수 있다.
biosemi_montage.plot(show_names=False, sphere=0.07)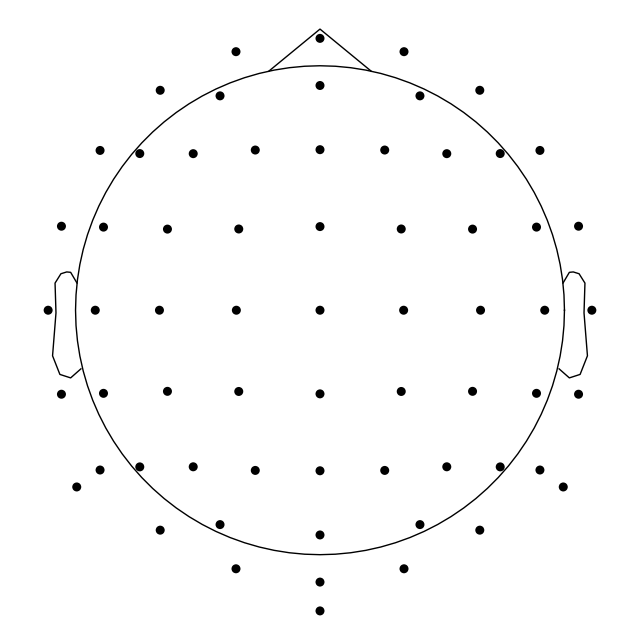
biosemi_montage.plot(show_names=False, sphere=(0.03, 0.02, 0.01, 0.075))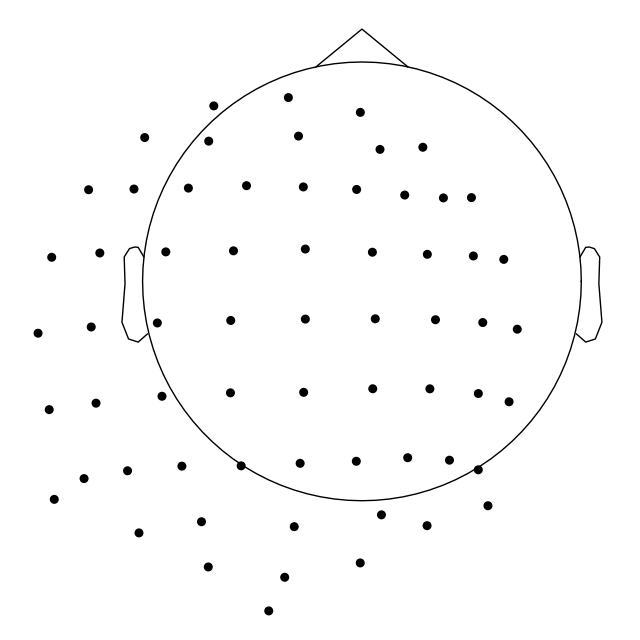
MNE-Python에서 머리 중심과 구 중심은 기준점을 사용하여 계산된다. 이것은 머리 원이 nasion 및 귀 높이에서 머리 둘레를 나타내며 10 - 20 EEG 시스템 (T4 / T8, T3 / T7, Oz 및 Fz에서 nasion 위)에서 일반적으로 측정되는 곳이 아님을 의미한다. 아래에서 기본적으로 T7과 Oz는 머리 원 안에 배치된다.
biosemi_montage.plot()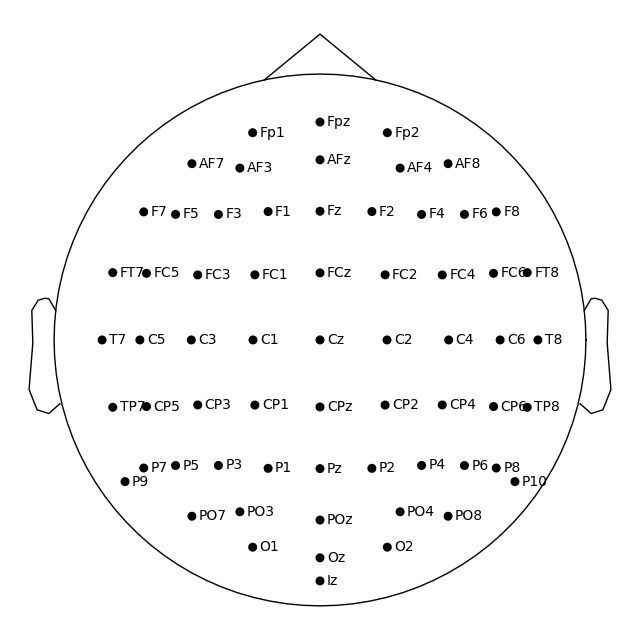
10 – 20 규칙 (EEGLAB에서도 사용됨)을 사용하여 머리 원을 그리는 것을 선호하는 경우 z 차원을 따라 구 원점을 몇 센티미터 위로 이동할 수 있다.
biosemi_montage.plot(sphere=(0, 0, 0.035, 0.094))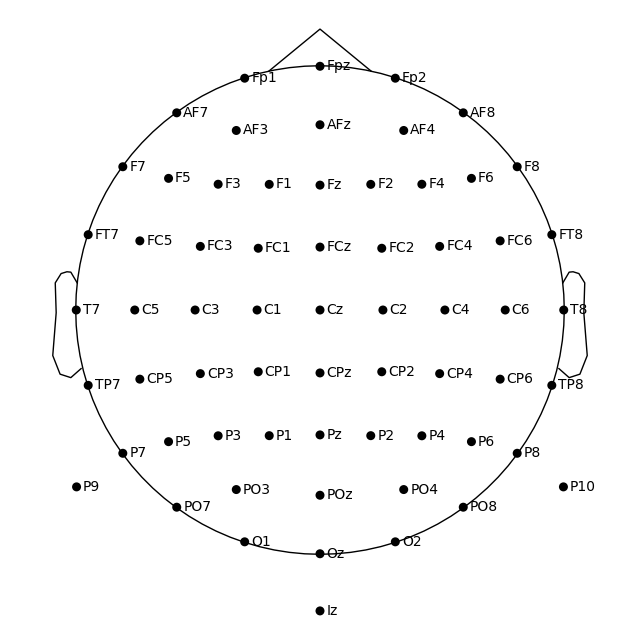
또는 Oz, Fpz, T3 / T7 또는 T4 / T8 채널에서 구 원점을 계산할 수 있다. 몽타주가 데이터에 적용되고 채널 위치가 헤드 스페이스에 있으면 이 작업이 더 쉽다 (예 참조).
https://mne.tools/stable/auto_tutorials/intro/40_sensor_locations.html
Working with sensor locations — MNE 1.0.0 documentation
In the sample data, the sensor positions are already available in the info attribute of the Raw object (see the documentation of the reading functions and set_montage for details on how that works). Therefore, we can plot sensor locations directly from the
mne.tools
'Brain Engineering > MNE' 카테고리의 다른 글
| [MNE-Python] 구성 (1) (0) | 2022.03.23 |
|---|---|
| [MNE-Python] 센서 위치 작업 (2) (0) | 2022.03.23 |
| [MNE-Python] 정보 데이터 구조 (0) | 2022.03.23 |
| [MNE-Python] 원시 데이터에서 이벤트 구문 분석 (2) (0) | 2022.03.23 |
| [MNE-Python] 원시 데이터에서 이벤트 구문 분석 (1) (0) | 2022.03.23 |



